PrimerPlex - 多重PCR引物设计软件
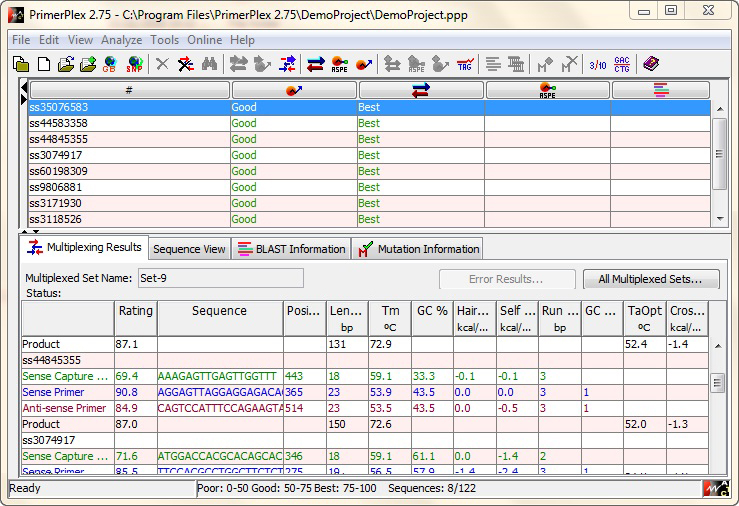
PrimerPlex是一种高效且复杂的工具,用于设计多重PCR的引物。多重分析有助于在单个反应容器中扩增多个目标,从而减少试验时间和成本。
用于多重PCR和高通量SNP基因分型和分析的寡核苷酸设计
多重PCR的引物设计存在若干挑战,包括引物二聚体、无法分离具有相似电泳迁移率的扩增子以及因与非目标DNA模板的非特异性结合而导致的错误引物。PrimerPlex采用专有算法,在统一的反应条件下为100多个序列设计理想的多重引物组。引物组是在筛选了池中的全部引物并尽量减少Tm错配后确定的,以确保特定的扩增和高信号强度。在这个过程中,它在几秒钟内分析了数百万个可能的多重引物集,并提出了一个备用集的清单。你可以指定所设计的引物对之间的minimum产物大小差异,以便在凝胶上更好地显示条带。为了保证引物的特异性,引物可以从PrimerPlex与NCBI的任意基因组数据库进行BLAST搜索。
对于SNP基因分型研究,PrimerPlex设计了SNP侧翼引物。结果包括每个DNA模板的引物对,包括产物大小、退火温度和其他物理化学寡核苷酸特性。二级结构的图形显示也可以帮助确定它们是否会阻碍反应。
多重PCR检测用于基因表达分析(使用凝胶电泳的终点检测)、高通量SNP应用(例如基因分型)、下一代测序所需的目标富集、病原体检测、菌株分型和单倍体分型。PrimerPlex还为高通量SNP基因分型分析设计了理想等位基因特异性捕获探针和等位基因特异性引物延伸 (ASPE) 引物。
PrimerPlex特征:
多重SNP检测和高通量基因分型
使用PrimerPlex,您可以设计SNP侧翼的多重PCR引物、3' 端具有SNP的ASPE引物以及捕获中心具有SNP的探针。PrimerPlex使您能够通过为每个突变设计寡核苷酸来检测中等大小基因组序列的全部突变。检查这些引物和探针的多重性,并显示可能的理想集合。
多重PCR引物设计
设计适用于多重检测的引物组可能是一项具有挑战性的任务。PrimerPlex使用专有算法来设计多重引物组,很大限度地减少 Tm错配。分析全部组的交叉反应性以确保特异性和有效扩增。您可以从以各种可排序顺序呈现的替代列表中选择适合您的实验需求的集合。使用PrimerPlex,您可以灵活地设计引物的3'末端、5'末端、距这些末端的指定距离或序列内的任意位置。
对于SNP基因分型分析,该程序设计侧翼突变的引物,例如SNP、DIP(缺失/插入多态性)和MNP(多核苷酸多态性)。
检测支持
使用PrimerPlex,设计用于多重PCR、ASPE和直接杂交检测的寡核苷酸。筛选寡核苷酸的交叉反应性,以确保高信号强度。很大限度的减少Tm错配。筛选寡核苷酸的二聚体、运行和重复。
下一代测序分析
使用PrimePlex,设计用于预测序样品制备和突变检测的多重寡核苷酸,例如SNP、DIP(删除/插入多态性)和MNP(多核苷酸多态性)就像单击几个按钮一样简单。
引物特异性
程序BLAST在一次搜索运行中搜索多个序列。在引物设计期间会自动解释结果并避免识别出的同源性,从而确保设计具有高度特异性。
评估预先设计的引物和探针
PrimerPlex可以使用预先设计好的经过验证的寡核苷酸来构建多重集。在为每个序列指定寡核苷酸后,分析它们的特性,并提醒用户发现任意偏差。对于预先设计的寡核苷酸不可用的序列,PrimerPlex对其进行设计,检查它们的多路复用并突出显示兼容性问题(如果有)。然后,用户可以决定接受该设计或创建一个单独的池。此功能将控制权交给用户。
添加MagPlex TAG
该程序现在还支持MagPlex TAGs数据库以及MicroPlex xTAGs(以前称为FlexMAP TAGs),以便为每个ASPE引物自动添加适当的标签。这些标签的选择使得它们可以很大限度的减少二聚化。并且不会在它们所连接的寡核苷酸上折叠起来。此外,用户可以覆盖程序的推荐并自行选择标签。如果标签不是unique或者选择的标签与寡核苷酸不兼容,PrimerPlex会提醒用户。
系统要求:
| 对于Windows | 需要 | 推荐 |
| CPU | Pentium IV 1.80 GHz | Intel Core i3 (3rd Gen) |
| 内存 | 1GB可用RAM | 2GB或更高的可用RAM |
| 硬盘空间 | 500MB可用硬盘 | 1GB可用硬盘 |
| 屏幕分辨率 | 800×600 | 1024×768 |
Windows支持的平台:XP/Vista/Windows 7/Windows 8/Windows 10
【英文介绍】
Oligo Design for Multiplex PCR & High Throughput SNP Genotyping and Analysis
PrimerPlex is an efficient and sophisticated tool for designing oligos for multiplex assays. Multiplex assays facilitate amplification of multiple targets in a single reaction vessel, reducing both the time and cost of experimentation.
Primer design for multiplex PCR presents several challenges which include primer dimers, inability to separate amplicons with similar electrophoretic mobility and mis-priming due to nonspecific binding to non-target DNA templates. PrimerPlex uses proprietary algorithms to design optimal multiplex primer sets under uniform reaction conditions for over 100 sequences. The primer sets are identified after screening all the primers in a pool and minimizing Tm mismatches to ensure specific amplification and high signal strength. In this process, it analyzes millions of possible multiplex primer sets in a few seconds and presents a list of alternate sets. You can specify minimum product size differences amongst the set of designed primer pairs for better visualization of bands on a gel. To assure primer specificity, primers can be BLAST searched from PrimerPlex against any of the genomic databases available at NCBI.
For SNP genotyping studies, PrimerPlex designs SNP flanking primers. The result includes a primer pair for each DNA template with product size, annealing temperature and other physio-chemical oligo properties. A graphical display of secondary structures is also available to help ascertain if they would impede the reaction.
The multiplex PCR assays are used for gene expression analysis (end point detection using gel electrophoresis), for high throughput SNP applications such as genotyping, for target enrichment required in Next Generation Sequencing, pathogen detection, strain typing, and haplotyping. PrimerPlex additionally designs optimal allele specific capture probes and Allele Specific Primer Extension (ASPE) primers for high throughput SNP genotyping assays.
- 2026-02-09
- 2026-01-20
- 2026-01-16
- 2026-01-12
- 2026-01-12
- 2026-01-09
- 2026-03-09
- 2026-03-09
- 2026-03-09
- 2026-03-09
- 2026-02-26
- 2026-02-26



















