AlleleID - 物种识别和分类鉴定软件
AlleleID是一款综合性桌面工具,旨在应对细菌鉴定、病原体检测或物种鉴定的挑战。以ClustalW多序列比对为核心,AlleleID可用于设计用于微阵列或实时PCR的物种鉴定/跨物种探针,包括SYBR® Green、TaqMan® MGB、TaqMan® probes、Molecular Beacons和实时PCR引物。AlleleID还支持设计用于检测可变剪接事件的微阵列实验。

物种鉴定试验/跨物种试验/AlleleID鉴定试验
AlleleID使用ClustalW比对序列并分析保守和物种特异性区域。然后,您可以使用该程序进行实时PCR引物设计(包括SYBR® Green引物设计)和双标记探针设计(TaqMan® probes、TaqMan® MGB probes和molecular beacons)。这些测定旨在只检测混合物中的菌株(菌株检测)或感兴趣的物种。
用于检测成功的复杂算法
通过避免通过自动解释BLAST搜索结果发现的同源区域来设计高度特异性的寡核苷酸。通过避免模板二级结构来提高实时PCR引物和探针的效率。“Minimal Set”是该程序中具有的功能之一,可帮助设计少量的allele特异性寡核苷酸引物和双标记探针。从混合物中识别每个所需的物种/菌株/分类群,从而降低分析成本。对于分类群或跨物种分析中,当组或分类群高度不同时,此功能很有用。对于部分预先设计的、经过验证的引物组,AlleleID®可以为其余序列设计兼容的引物和探针,用于物种鉴定或分类特异性分析。
广泛支持实时或定量PCR检测和SNP/表达微阵列
AlleleID设计了优化的SYBR® Green引物、TaqMan® probes、TaqMan Minor Groove Binding(MGB)probes、FRET probes或molecular beacons,用于实时定量PCR差异基因表达和SNP基因分型分析。您还可以在一次运行中设计多达一万个序列的实时PCR引物和双标记探针,用于制作SNP检测或表达微阵列。
设计Luminex xMAP®检测
AlleleID®为基于Luminex的xMAP®技术的悬浮阵列系统设计菌株分化多重分析。如果您正在使用密切相关的生物体,此功能将使您能够在单个反应容器中运行Allele特异性引物延伸(ASPE)和DHA测定。
用于拷贝数和突变检测的多重连接依赖性探针扩增(MLPA)测定
AlleleID®是一个为MLPA或MRC Holland引入的多重连接依赖性探针扩增技术设计合成探针的程序,使研究人员能够在没有合适的试剂盒时扩展该技术。它是一种快速发展的高通量遗传分析方法。它可以检测外显子缺失、拷贝数变化和CpG甲基化模式。它具有成本效益、灵敏、特异性和可重复性。
AlleleID®还为PamGene Arrays设计了mRNA特异性、DNA特异性和突变特异性MLPA探针。这将使PamChip®用户能够设计自己的探针,以使用PamGene的高精度检测平台检测感兴趣的基因。结合这两种技术,只需6小时即可轻松检测到拷贝数变化和突变。
AlleleID特点:
用于跨物种和分类特定应用的实时PCR和微阵列引物/探针设计
-
对于跨物种分析,AlleleID®识别保守区域以设计通用探针。在设计诊断分析时,此功能对于检测物种或分类群很有用。当所研究的生物体的基因组草图不可用时,此功能还可用于帮助研究基因表达。
-
当无法识别一组序列的紧要保守区域时,AlleleID®会尝试设计“Mismatch Tolerant”probe。probe可以包括至多指定数量的错配碱基,这些错配碱基尽可能靠近5'端。
-
还包括“Minimal Set”选项,可帮助您设计少数量的探针组,以识别序列,从而降低总体分析成本。
跨物种检测
在进化过程中,生物体DNA组成的变化,例如单核苷酸多态性,导致了表型性状的多样性。一代又一代地继承这些变化并为物种的生物多样性做出贡献。尽管发生了全部这些变化,但仍有一段DNA在进化过程中保持保守。此类保守区域或序列可用于提示系统发育相似的群体、识别群体内的生物或了解其在特定区域的植物群中的生物多样性程度。
现代跨物种检测技术
实时PCR和微阵列为诊断病原体提供了一种快速、灵敏、特异性和定量的工具。当样本中存在不止一种病原体时,疾病不轻易根据症状来区分。显微镜鉴定需要在专门的培养基上培养数天至数周。近来,实时PCR检测已成功用于检测真菌病原体,如Rhizoctonia, Pythium, and Fusarium。
设计跨物种分析涉及使用多序列比对工具来识别DNA的保守片段,以及设计用于检测它的杂交探针。近来,使用分子水平鉴定技术(如实时PCR的微阵列)在分类水平上对微生物进行鉴定取得了进展。
AlleleID®如何设计跨物种/类群特异性分析
AlleleID®设计了实时PCR分析,包括molecular beacons、TaqMan® probes、TaqMan® MGB和SYBR® Green引物,以及用于设计用于分类区分的通用探针的微阵列。
在设计分类群特异性分析时,AlleleID®设计了一个通用探针来识别混合物中的一个分类群或一组生物。当此类设计不可行时,对于具有挑战性的问题,AlleleID®尝试通过很大限度地减少错配来进行设计,尤其是在问题较多的 3' 端避免它们。

要设计特定分类/跨物种探针,请对齐序列或加载对齐文件
AlleleID®为设计特定分类群/跨物种分析提供了多种设计选项
带有保守引物的保守探针: AlleleID®通过比对序列来识别保守区域,然后在其中设计探针。这使得能够用单个探针识别集合中的任意序列。它还在探针两侧的保守区域设计引物对。因此,单个引物对可以扩增一个组或一个分类群的全部序列。

类群特异性/跨物种鉴定:具有保守引物的保守探针
类群特异性/跨物种鉴定:具有理想引物的保守探针
Conserved Probe with Optimal Primers: AlleleID®设计了一个引物对,可以在比对中扩增尽可能多的序列。设计引物有两种选择,Unique和Optimal。对于“Unique”,程序设计了一个引物对来扩增每个序列。每个引物与其模板同源。对于“Optimal”,该程序很大限度地减少了扩增全部序列所需的引物数量;利用引物可以与序列结合并扩增它的事实,即使它们包含有限的不匹配的碱基,特别是在引物的5'端。
类群特异性/跨物种鉴定:具有不匹配碱基和保守引物的探针
Probes with Mismatched Bases and Conserved/Optimal Primers: AlleleID®可以设计具有特定数量错配碱基的探针。不匹配的碱基将遵循多数共识,这意味着探针将具有比对中常见的碱基。这种设计很大限度地降低了成本,这是一个关键的考虑因素,尤其是在诊断分析设计中。引物设计在保守区域。为了降低分析成本,它设计了在比对中扩增保守区域所需的少量的引物对。
类群特异性/跨物种鉴定:具有不匹配碱基和理想引物的探针
Minimal Probes with Optimal Primers: 当比对序列之间的同源性很低且上述两种设计(单个分类群特异性探针组或具有错配耐受性的探针组)都不可行时,AlleleID®设计尽可能少的探针以检测比对中的全部序列。该程序先设计对多数共识的探针,然后对少数共识和剩余的单个序列进行探针设计。这种设计很有用,特别是对于研究从不同地理位置获得的序列之间的系统发育关系。
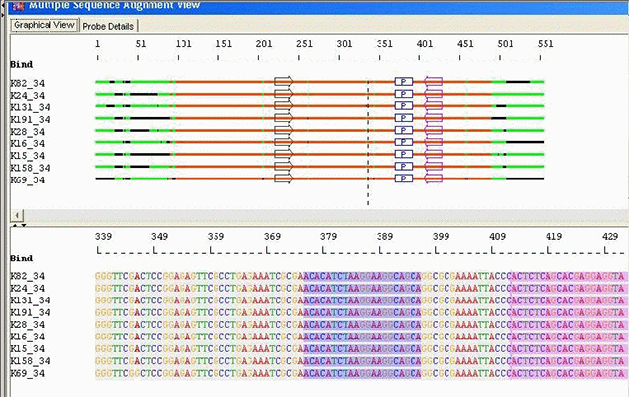
类群特异性/跨物种鉴定:具有错配碱基和保守引物的探针
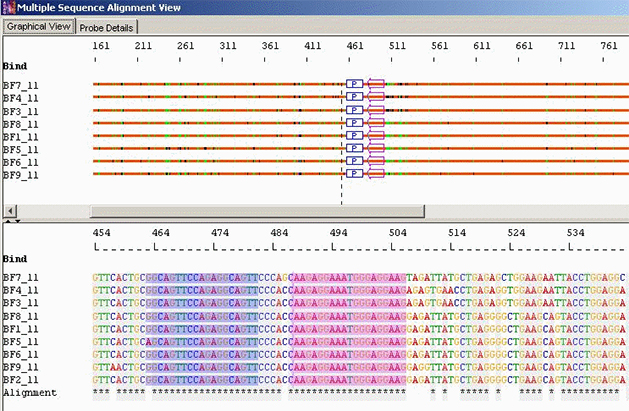
类群特异性/跨物种鉴定:具有错配碱基和理想引物的探针
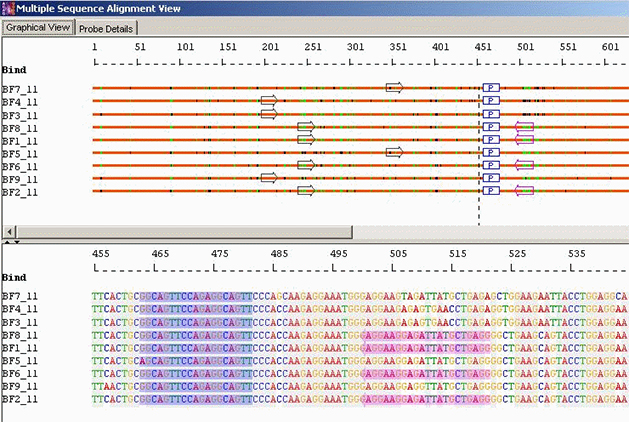
Minimal Probes with Optimal Primers: 当比对序列之间的同源性很低且上述两种设计(单个分类群特异性探针组或具有错配耐受性的探针组)都不可行时,AlleleID®设计尽可能少的探针以检测比对中的全部序列。该程序先设计对多数共识的探针,然后对少数共识和剩余的单个序列进行探针设计。这种设计很有用,特别是对于研究从不同地理位置获得的序列之间的系统发育关系。
类群特异性/跨物种鉴定:Minimal Probes with Optimal Primers
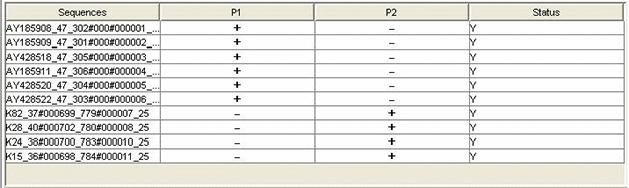
Y代表可以识别的序列
物种鉴定
使用AlleleID®,您可以设计用于从混合序列中识别物种和菌株的检测方法
-
指定序列:通过将它们移动到“Bind”框来指定要在混合中检测的序列。
-
引物设计的选择:引物设计有两种选择,Unique和Optimal。当您选择Unique时,程序会设计一个引物对来扩增每个序列。对于Optimal,该程序很大限度地减少了扩增全部序列所需的引物数量。目标是小化合成和总体分析成本。
-
探针设计的选择:与探针类似,当您选择Unique时,AlleleID®为每个序列设计一个探针。当您选择Minimal set选项时,AlleleID®设计了识别序列所需的少量的探针。
历史上如何识别物种
在疾病诊断和药物开发领域,用于细菌和物种鉴定的基于培养的方法已被更快、更可靠的基于分子的技术(如实时PCR和微阵列)迅速取代。这些技术成功的原因是人们可以很轻易地从从空气、土壤或水等复杂来源收集的样本中识别出一个物种。
基于分析的技术使用针对目标基因的特定区域设计的物种特异性探针来检测和鉴定细菌。微阵列可以在复杂的背景下同时快速检测和识别多种病原体。
现代物种鉴定技术
AlleleID®设计了实时PCR分析,包括molecular beacons、TaqMan® probes、TaqMan®MGB和SYBR®Green引物)和用于细菌鉴定、病原体检测和分类/物种鉴定的微阵列。AlleleID®使用快速准确的ClustalW算法来比对序列以识别物种特定区域。
物种识别:对齐序列并识别特有区域
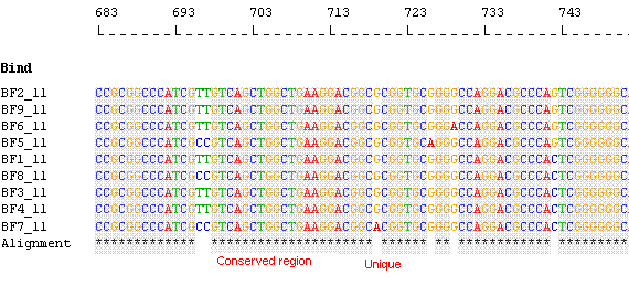
AlleleID®提供多种物种鉴定选项
-
Unique Probes with Unique Primers:当选择的探针类型为Unique时,AlleleID®设计探针来识别Bind To框中的每个序列,每个序列一个探针。程序会搜索排列中每个序列所特有的碱基位置。探针的中心尽可能靠近这些Unique的碱基,因此即使错配只一个碱基,检测也能成功。
物种鉴定:Unique Probes with Unique Primers
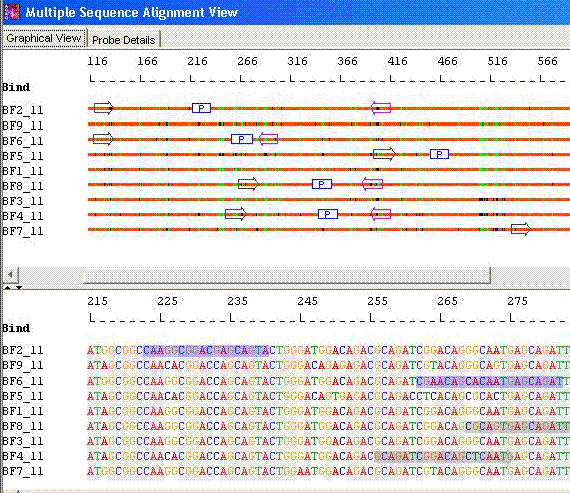
-
Minimal Set with Unique Primers: 选择Minimal set选项,AlleleID®设计了识别序列所需的少量的探针。Minimal set通常包含比要识别的序列数量更少的探针,并且探针的特有组合识别每个序列。这种测定的目的是使合成和总体测定成本小化。
物种鉴定:Minimal Set with Unique Primers
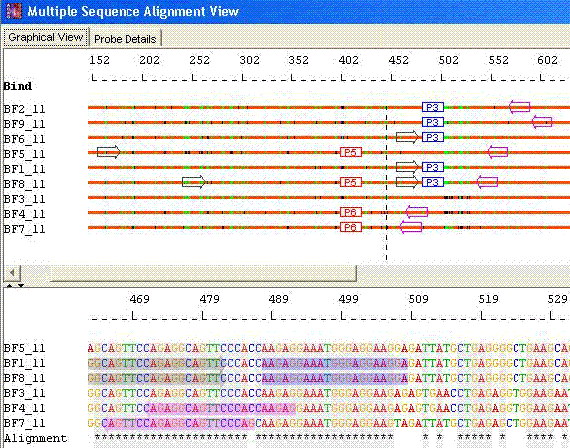
A "Minimal Probe" Matrix
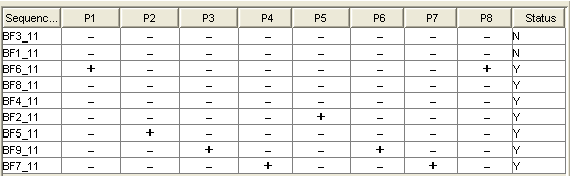
Y代表可以识别的序列。
-
Minimal Probes with Optimal Primers
对于Optimal,该程序很大限度地减少了扩增全部序列所需的引物数量。目标是小化合成和总体分析成本。
物种鉴定:Minimal probes with Optimal primers
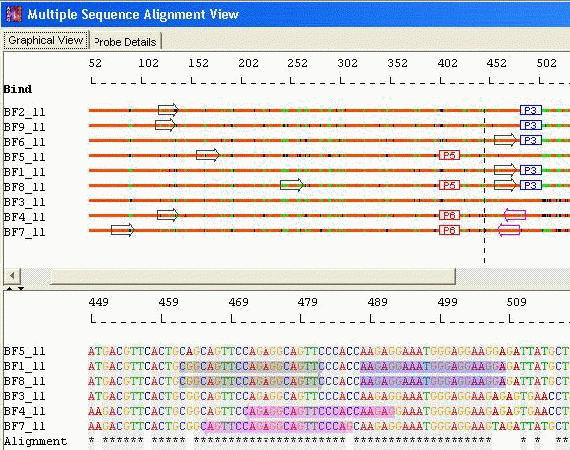
选择性拼接
DNA微阵列可能是剪接变异研究很有用的技术。AlleleID®包括设计用于检测可变剪接和基因剪接事件的微阵列探针的能力。
-
AlleleID®设计了两种类型的微阵列探针,我们称之为junction probes和exon probes,让用户可以控制设计参数。
-
AlleleID®通过为每个接头和/或创建的每个剪接变体设计探针来检测可变剪接事件。
-
AlleleID®设计exon probes。这种探针对于确认变体中是否存在exon很关键。
-
基因剪接技术资源。
什么是选择性剪接?
基因的编码和非编码片段可以以不同的方式排列。当这从同一亲本基因产生不同的m-RNA序列时,这种现象被称为可变剪接。这个过程的生物学后果可能很严重,因为相同的基因会导致形成不同的蛋白质,这些蛋白质可能是功能性的、非功能性的或功能障碍的。剪接事件的各种形式被称为剪接变体。如上所述,AlleleID®可用于使用微阵列实验进行剪接变异检测。
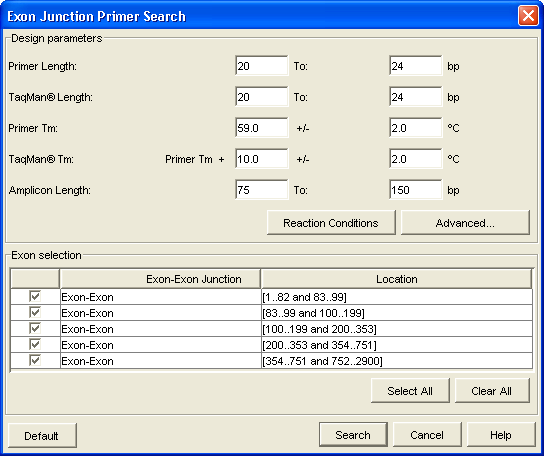
Exon Junction引物设计
使用AlleleID®,您还可以设计across exon junctions的引物。通过解释GenBank标头注释或手动指定它们来识别exon。
为了从cDNA和gDNA的混合物中选择性地扩增cDNA,设计引物使得一对引物中至少有一个引物across exon junctions。引物设计用于与由其合成的mRNA或cDNA退火,但不与基因组DNA退火。
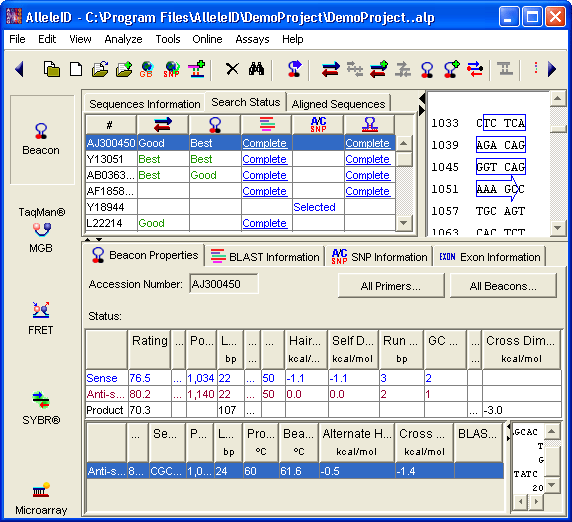
设计TaqMan® Probes
-
TaqMan® Probes:AlleleID®设计了理想的TaqMan® Probes,不含二聚体、重复和运行,以确保信号保真度。
-
TaqMan® MGB Probes:AlleleID®支持TaqMan® MGB Probes的设计,与常规TaqMan® Probes相比,该Probes通常更短。
-
多重和等位基因鉴定分析: AlleleID®支持多达四个序列的多重化,避免与全部双标记探针和引物的交叉同源性,防止多重反应中的竞争。设计用于SNP鉴定的野生和突变TaqMan® Probes。
-
评估预先设计的基于TaqMan® Probes的检测:可以评估预先设计的引物组并设计兼容的TaqMan® Probes。同样,可以评估预先设计的TaqMan® Probes并设计兼容的引物。您甚至可以导入在SYBR® Green引物设计模式中设计的引物,然后设计兼容的探针。
-
图形视图:显示TaqMan® Probes二级结构的图形视图。
-
排名:使用统计优化技术,只为每个模板设计好的TaqMan® Probes。

SYBR® Green Assays的引物设计
-
设计引物:设计针对SYBR®Green分析优化的引物。
-
避免同源性: AlleleID®通过BLAST搜索您的序列来进行高特异性的SYBR® Green引物设计,自动解释结果,然后设计高特异性的SYBR® Green引物。
-
BLAST: SYBR® Green引物可以针对NCBI提供的基因组数据库进行BLAST。这有助于验证SYBR® Green引物设计并可视化引物和扩增子的特异性。
-
模板二级结构:在设计SYBR® Green引物时避免识别模板结构。
-
算法:使用nearest neighbor热力学算法计算SYBR® Green Primer Tm。
-
SYBR® Green Primer Rating:根据预期的引物效率对设计的SYBR® Green引物进行排名。
-
综合标准:筛选SYBR® Green引物的热力学特性、位置和二级结构。
-
多重引物:避免与全部其他二聚体的交叉同源性以减少引物二聚体。
-
预先设计的引物:设计理想的TaqMan® Probes和molecular beacons,并与经过充分验证的SYBR® Green引物一起使用。此外,您可以使用预先设计或已发表或经过充分验证的正向或反向引物,并让AlleleID®设计其余的分析。例如,对于给定的正向引物,AlleleID®将为SYB®R Green检测设计反向引物,并为TaqMan®检测设计反向引物和TaqMan® Probes。
-
SNP扩增:设计SYBR® Green引物以扩增SNP位点。AlleleID®甚至可以分析SNP特异性引物,以帮助您确定它们对反应的适用性。

Molecular Beacon设计
-
Tm调整:自动选择适当长度的杆以获得理想信标Tm。
-
Optimal Beacons:设计没有二聚体、重复和运行的信标,以提高信号强度。
-
多重信标和引物:检查分子信标与全部引物的交叉同源性,防止多重反应中的竞争。
-
等位基因鉴定:设计用于检测野生和突变等位基因的分子信标。
-
Tm计算:高精度hairpin Tm计算。
-
评估预先设计的分子信标检测:可以评估预先设计的引物组或构建预先设计的分子探针的引物。类似地,可以评估预先设计的信标或可以为预先设计的引物组设计信标。这有助于将SYBR® Green引物用于信标测定,可用于细菌鉴定测定或任意其他测定。
-
闪电般的快速:在一秒钟内处理平均cDNA序列。
-
图形视图:以图形方式显示设计的信标及其属性。
FRET Probe设计
-
Optimal FRET Probes: 设计不含二聚体、重复和运行的Optimal FRET Probes,以确保信号保真度。
-
等位基因鉴定: AlleleID®设计了用于检测野生和突变等位基因的FRET Probes。
-
评估预先设计的FRET分析:可以评估预先设计的引物组或构建预先设计的探针的引物。您甚至可以导入在SYBR® Green引物设计模式中设计的引物。
-
图形视图:以图形方式显示设计的FRET probe及其属性。

高通量微阵列探针设计
-
寡核苷酸设计: AlleleID®设计用于SNP基因分型和表达研究的高度特异性寡核苷酸微阵列。
-
cDNA微阵列设计: AlleleID®使用优化的设计参数,帮助设计用于中等到大型扩增子的引物。
-
SNP检测:设计用于杂交和引物延伸检测的SNP probes。
-
多个probes:设计每个序列的best probe或每个序列的多个probes,以提高检测的准确性。
数据库和数据管理
项目:允许创建多个项目。通过为每个实验创建单独的项目,可以在AlleleID®中轻松组织和管理大量数据。
内置数据库: AlleleID®为序列信息和搜索结果维护一个本地数据库。
数据导出: AlleleID®以制表符分隔格式导出数据,以便轻松加载到电子表格和数据库中。
输入输出
-
生成报告:您可以为使用AlleleID®设计的分析创建格式精美的报告。它应该有助于记录保存和与同事共享信息。该报告有助于可视化引物和探针在序列上的位置,包括替代引物和探针的列表,显示引物、探针、扩增子和序列属性以及使用的设计参数。
-
序列详细信息视图:项目中全部序列的综合信息可使用内置数据库在本地获得,并且可以使用浏览器从程序内查看序列详细信息。
-
序列可视化: AlleleID®以图形方式显示序列上的引物和探针。
-
输入格式:支持标准GenBank和FASTA格式的序列。使用多重检索工具,可以从本地驱动器加载序列。
-
SNP加载:从标准GenBank变异文件中的变异描述符轻松加载数千个SNP。
-
在电子表格中查看输出:可以在任意电子表格(如MS Excel、Lotus 123或StarSuite电子表格)中查看和处理结果。
系统要求:
| 对于Windows | 需要 | 推荐 |
| CPU | Pentium IV 1.80 GHz | Intel Core i3 (3rd Gen) |
| 内存 | 1GB可用RAM | 2GB或更高的可用RAM |
| 硬盘空间 | 500MB可用硬盘 | 1GB可用硬盘 |
| 屏幕分辨率 | 800×600 | 1024×768 |
Windows支持的平台:XP/Vista/Windows 7/Windows 8/Windows 10
【英文介绍】
AlleleID is a comprehensive desktop tool designed to address the challenges of bacterial identification, pathogen detection or species identification. With ClustalW multiple sequence alignment at its core, AlleleID can be used to design species identification/cross species probes for microarrays or real time PCR including SYBR Green, TaqMan MGB, TaqMan probes, Molecular Beacons and real time PCR primers. AlleleID also offers support for designing microarray experiments for detecting alternative splicing events.
Species Identification Assays/Cross Species Assays/Allele Identification Assays
AlleleID aligns sequences using ClustalW and analyzes conserved and species specific regions. You can then use the program for real time PCR primer design (SYBR Green primer design included) and dual labeled probe design (TaqMan probes, TaqMan MGB probes and molecular beacons). These assays are designed to detect only the strain (strain detection) or species of interest from the mix.
For cross species assays, AlleleID identifies the conserved regions to design a universal probe. For related organisms, AlleleID can be used to study gene expression when genome draft of the organism under study is not available. This powerful functionality is sure be helpful for many challenging tasks such as detection, identification, quantification or monitoring of contaminants, environments...
Sophisticated Algorithms for Assay Success
Highly specific oligos are designed by avoiding regions of significant homologies found by automatically interpreting BLAST search results. Real time PCR primer & probe efficiency is enhanced by avoiding template secondary structures. "Minimal Set", one of the most innovative features in the program, helps design the fewest number of allele specific oligonucleotide primers and dual labeled probes that uniquely identify each of the desired species/strain/taxa from the mix, lowering assay costs. For taxa or cross species assays, this feature is especially useful when the group or taxa is highly dissimilar. For a partial set of pre-designed, proven set of primers, AlleleID can design compatible primers and probes for the rest of sequences for species identification or taxa specific assays.
Extensive Support for Real Time or Quantitative PCR Assays & SNP/Expression Microarrays
AlleleID designs optimal SYBR Green primers, TaqMan probes, TaqMan Minor Groove Binding (MGB) probes, FRET probes or molecular beacons for real time quantitative PCR differential gene expression and SNP genotyping assays. You can also design real time PCR primers and dual labeled probes for up to ten thousand sequences in a single run for making SNP detection or expression microarrays.
Design Luminex xMAP Assays
AlleleID designs strain differentiation multiplex assays for Suspension Array systems based on Luminex's xMAP technology. If you are working with closely related organisms, this functionality will enable you to run Allele Specific Primer Extension (ASPE) and DHA assays in a single reaction vessel.
Multiplex Ligation-dependent Probe Amplification (MLPA) Assays for Copy Number and Mutation Detection
AlleleID is the only program that designs synthetic probes for MLPA or Multiplex Ligation-dependent Probe Amplification technique introduced by MRC Holland enabling the researcher to extend the technique when appropriate kits are unavailable. It is a rapidly growing high throughput method for genetic profiling. It can detect exon deletions, copy number changes, and CpG methylation patterns. It is cost-effective, sensitive, specific and reproducible.
AlleleID also designs mRNA specific, DNA specific and mutation specific MLPA probes for PamGene Arrays. This will enable PamChip users designing their own probes to detect genes of interest using PamGene's high precision detection platform. Combining these two technologies, copy number changes and mutation can be easily detected in just 6 hours.
- 2026-01-20
- 2026-01-16
- 2026-01-12
- 2026-01-12
- 2026-01-09
- 2026-01-05
- 2026-02-05
- 2026-02-05
- 2026-01-28
- 2026-01-26
- 2026-01-26
- 2026-01-16


















